- Instalación de prerrequisitos
- Instalación de Spark
- Instalación de R y SparkR
- Gestionar los nodos del cluster
- Trabajar con SparkR
R es una magnífica herramienta para llevar a cabo todo tipo de tareas de análisis exploratorio de datos de manera interactiva, desde una consola en la que introducimos órdenes más o menos simples y obtenemos resultados, en forma de tablas, indicadores estadísticos o gráficas, de manera inmediata. El enorme número de paquetes disponibles simplifica nuestro trabajo, y cualquier secuencia de operaciones puede convertirse fácilmente en un script que permite reproducir los experimentos tantas veces como sea necesario sobre distintos conjuntos de datos.
La mayor limitación de R es que solamente podremos usar los recursos (potencia de proceso y memoria) de la máquina en la que estamos trabajando, algo que dificulta el trabajo con grandes volúmenes de datos (el repetido hasta la extenuación big data). En el artículo Configuración de un cluster con Ubuntu y SGE expliqué cómo configurar un cluster de máquinas GNU/Linux y un sistema de gestión de colas. Con dicha configuración es posible distribuir un lote de tareas, escritas como scripts R, entre las distintas máquinas del cluster, reduciendo el tiempo global. No obstante, cada tarea estará limitada por los recursos de la máquina en que se ejecuta, además de ser un enfoque no adecuado para el uso interactivo sino la ejecución de trabajos por lotes.
Una alternativa al uso de colas, ejecutando tareas independientes en cada una de las máquinas del cluster, es el uso de plataformas como Apache Hadoop, que implementan un sistema de archivos distribuido (HDFS) y un mecanismo de procesamiento en paralelo (Map/Reduce) capaz de dividir una tarea en tantas porciones como sea necesario, distribuirlas entre las máquinas del cluster y después componer el resultado final a partir de los resultados parciales que van obteniéndose. El paradigma Map/Reduce no deja de ser un enfoque de bajo nivel a un problema, el aprovechamiento de los recursos distribuidos, y desde luego no resulta lo más adecuado para un uso interactivo. Además el esquema de funcionamiento depende fuertemente de la escritura en disco de porciones de datos y resultados parciales, lo cual conlleva un cierto nivel de carga sobre el sistema de entrada/salida de las máquinas, ralentizando la consecución del objetivo final.
Frente a Hadoop, Apache Spark representa una alternativa de más alto nivel para computación distribuida, pasando del enfoque Map/Reduce al uso de estructuras de paralelismo que resultan más naturales para el desarrollador. Spark no requiere escritura en disco de porciones de datos y resultados parciales, potenciando en su lugar el mayor uso de la memoria disponible en cada máquina del cluster. El resultado es que una misma solución puede ejecutarse hasta 100 veces más rápido sobre Spark que sobre Hadoop. Además Spark ofrece una línea de comandos, en principio para los lenguajes Scala y Python, que permite operar sobre el cluster de manera interactiva.
Aunque durante un tiempo utilicé Python como lenguaje para el análisis exploratorio de datos y algunas tareas de minería de datos, desde hace años R es mi herramienta fundamental para estas tareas. Gracias al paquete SparkR, que actúa como puente entre Spark y R, es posible trabajar de manera interactiva aprovechando los recursos de un cluster sin abandonar la comodidad de la línea de comandos de R. En el resto de este artículo cuento cuáles son los pasos que he seguido para poder trabajar desde R sobre un cluster con Spark, partiendo de que tengo un conjunto de máquinas conectadas en red, tal y como explicaba en Configuración de un cluster con Ubuntu y SGE, y almacenamiento compartido NFS (otra alternativa será instalar Spark sobre Hadoop).
Instalación de prerrequisitos
Partiendo de una instalación de Ubuntu 14.04LTS, el primer paso ha sido instalar los prerrequisitos que necesita Spark para su funcionamiento. Entre estos se encuentran Java 7, Scala, Scala sbt y Python. El primer paso será añadir el repositorio desde el que instalaremos Java y la actualización de la base de datos. En Ubuntu recurriremos aadd-apt-repository y apt-get, en otras distribuciones, como CentOS, usaríamos yum, pero el procedimiento sería similar.
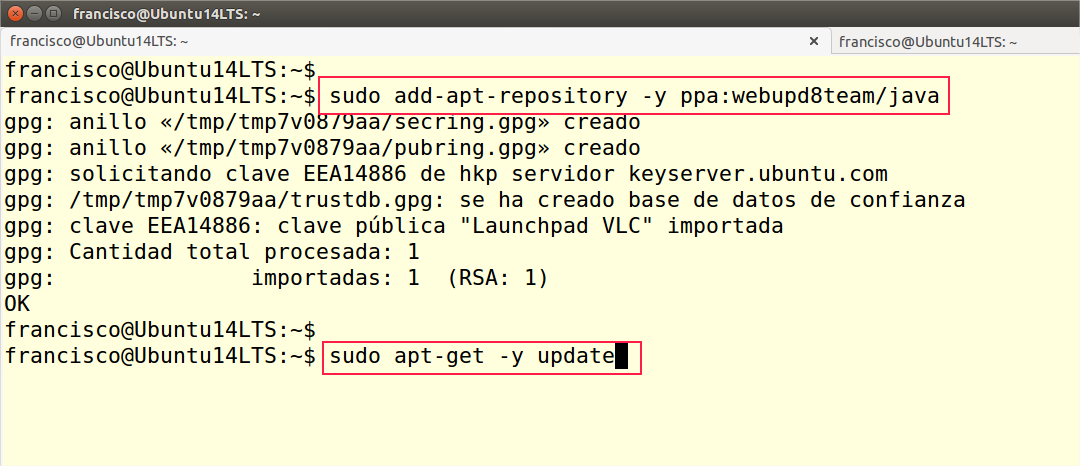
Actualizada la base de datos, usaríamos la siguiente secuencia de órdenes para instalar Java y Python junto con sus librerías:
sudo apt-get -y install oracle-java7-installer
sudo apt-get -y install python-software-properties
sudo apt-get -y install python-numpy python-scipy python-matplotlib ipython ipython-notebook
python-pandas python-sympy python-noseA continuación iremos al repositorio de Scala, descargaremos el paquete .deb más reciente, lo abriremos con el gestor de paquetes y procederemos a instalarlo, tal y como se aprecia en la siguiente imagen:
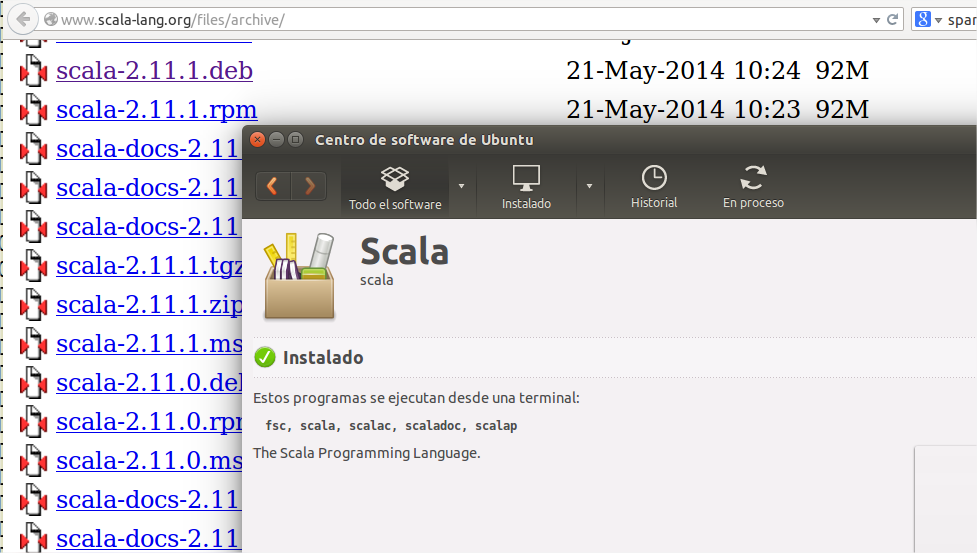
El siguiente software a instalar es Scala sbt, una herramienta que nos permitirá construir Spark a partir de su código fuente. Podemos descargarlo e instalarlo con el gestor de paquetes o bien hacerlo desde la línea de comandos, de la siguiente forma:
wget https://dl.bintray.com/sbt/debian/sbt-0.13.7.deb
sudo dpkg -i sbt-0.13.7.deb
sudo apt-get -y update
sudo apt-get -y install sbtTerminada esta última parte del proceso, ya tenemos todos los prerrequisitos necesarios para proceder a instalar Spark en nuestra máquina.
Instalación de Spark
En la web de Apache Spark podemos encontrar versiones precompiladas preparadas para funcionar sobre distintas versiones de Hadoop y otras plataformas software para procesamiento distribuido. En mi opinión lo recomendable (es lo que yo he hecho) es descargar el paquete de código fuente, configurar las variables necesarias para indicar cuál es nuestra versión de Hadoop y compilar Spark, obteniendo una versión a medida para nuestro sistema.
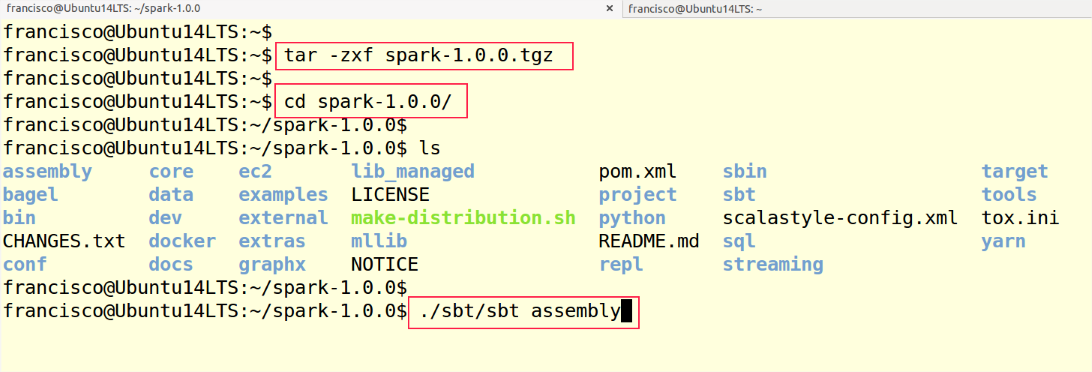
Una vez descargado el paquete que contiene el código fuente, procedemos a desempaquetarlo, entramos en el directorio donde se ha desempaquetado y procedemos a su compilación, pasos que quedan reflejados en la siguiente imagen. En caso de que necesitemos ajustar la versión de Hadoop, lo haríamos antes de proceder a compilar Spark con la última orden, siendo las indicaciones de la documentación oficial. También es posible instalar Spark sobre un cluster de ordenadores básico, sin Hadoop ni otro software de procesamiento distribuido. Es lo que yo he hecho, usando una unidad NFS como almacenamiento compartido en lugar del almacenamiento distribuido de Hadoop.
Finalizada la compilación de Spark, podemos comprobar que funciona adecuadamente iniciando su línea de comandos. Para ello usaremos la orden ./bin/spark-shell --master local. Tras el proceso de inicialización, durante el que se ponen en marcha distintos servicios incluido un servidor HTTP, deberíamos encontrarnos con una línea de comandos como la que aparece en la siguiente imagen.
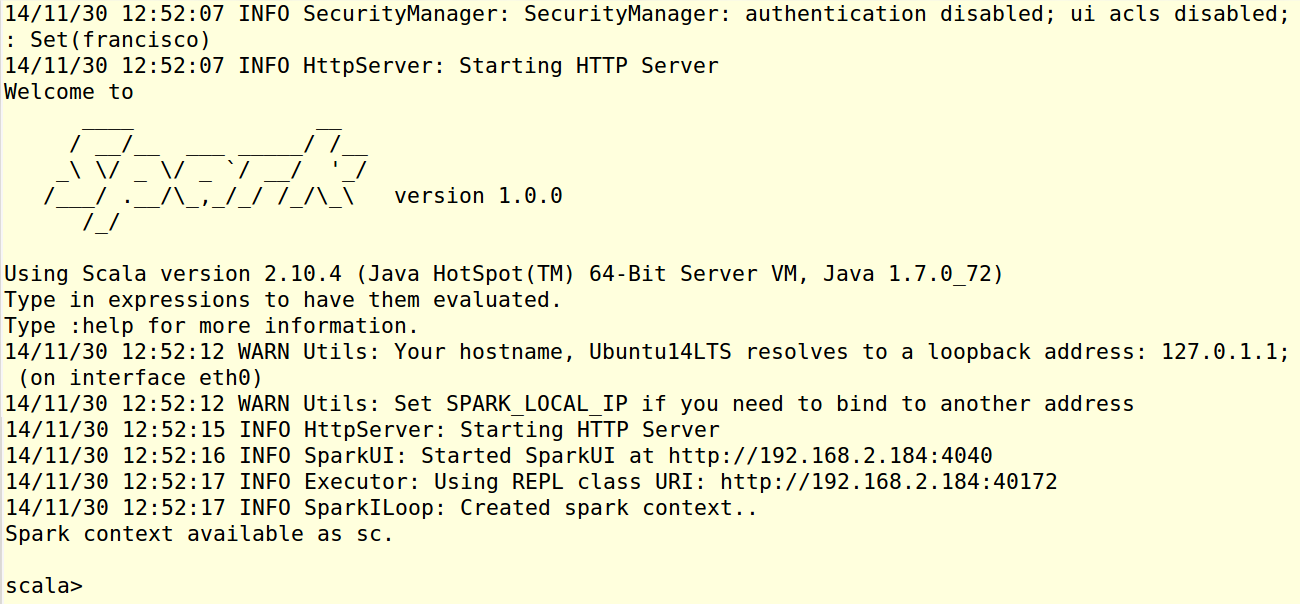
En este momento estaríamos usando la máquina local, en la que acabamos de hacer la instalación, como maestro del cluster. Por ahora es la única máquina en la que tenemos instalado Spark, pero no tendríamos más que clonar la máquina actual en otras, o repetir en cada una todo el procedimiento de instalación descrito hasta ahora, para tener múltiples máquinas con Spark, una actuando como maestro y el resto como esclavos. Antes de proceder a clonar, sin embargo, debemos completar la instalación y configuración de R y SparkR, de forma que ya estén en funcionamiento en los nodos esclavos.
Instalación de R y SparkR
Para instalar R en Ubuntu no tenemos más que ejecutar sudo apt-get install r-base r-base-dev desde la línea de comandos y esperar a que se descargue e instale el software. Tras esto debemos poder acceder a la consola de R simplemente escribiendo R en la línea de comandos de Bash. Una vez en la consola de R, procederemos a instalar el paquete rJava, necesario para poder instalar SparkR. Con este fin usaremos la orden install.packages("rJava"), tal y como se aprecia en la imagen inferior.
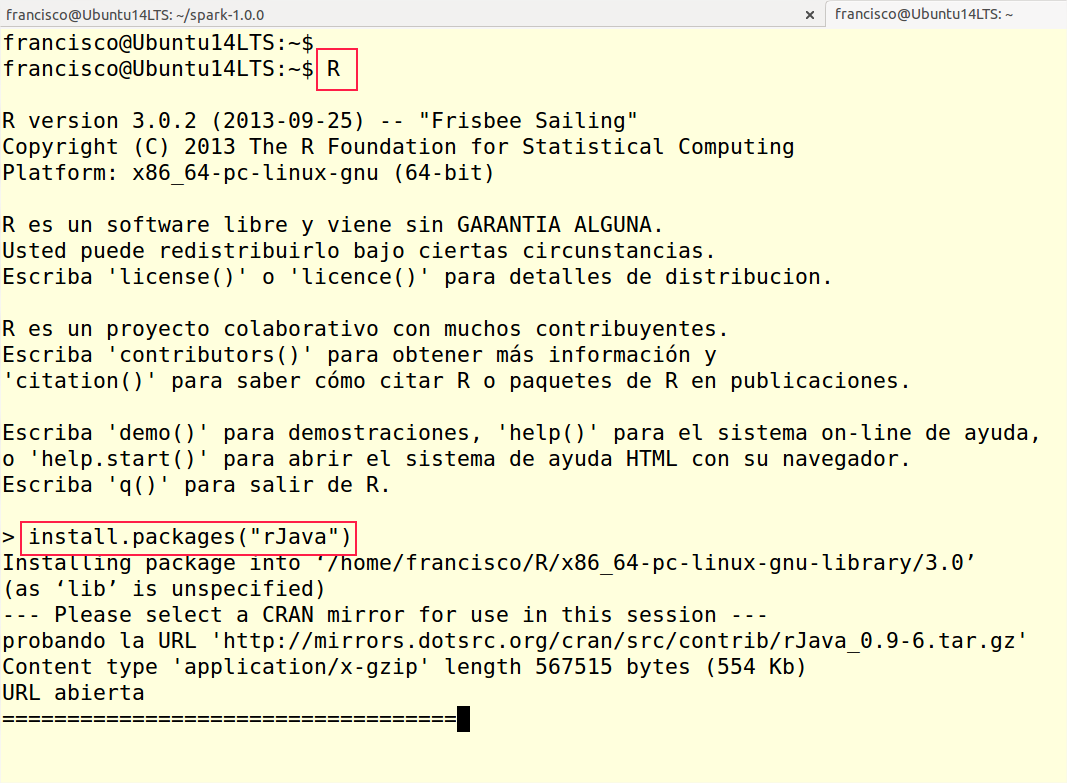
Tras salir de la consola de R (con la orden quit()), procedemos a descargar el paquete SparkR y a desempaquetarlo en un directorio. Entramos en él y ejecutamos ./install-dev.sh para lanzar la generación e instalación del paquete. Este consta de dos partes: un paquete R y un paquete Scala. Gracias a ellos, que actúan como pasarela, podremos acceder desde R a los servicios de SparkR. Terminado el proceso podemos usar la orden ./sparkR a fin de comprobar que podemos acceder a la consola de R con un contexto Spark ya disponible.
Gestionar los nodos del cluster
Finalizado todo el proceso de instalación y configuración del software, ahora sí estamos en disposición de clonar la máquina en el resto de equipos o bien repetir todo el proceso en cada una de ellas. La máquina cuya IP indique la variableMASTER actuará como maestra y todas las demás como esclavas. Si no se establece la mencionada variable, lo habitual es que el nodo maestro sea aquél desde el que gestionamos el cluster, máquina desde la que usaremos los scripts start-master.sh, start-slaves.sh, stop-master.sh y stop-slaves.sh descritos en la documentación de Spark para poner en marcha o detener tanto el maestro como los esclavos.
Para que el nodo maestro pueda conectar con los esclavos y lanzar trabajos es imprescindible que todos ellos sean accesibles mediante SSH desde el nodo maestro, usando una clave privada y sin contraseña. Las direcciones IP de los esclavos se facilitarán en el archivo conf/slaves. En caso de que necesitemos configurar aspectos como los puertos, la memoria a usar en cada nodo, etc., editaremos el archivo de configuración conf/spark-env.sh. En ambos casos conf es un subdirectorio que encontraremos en el directorio donde hayamos instalado Spark.
Trabajar con SparkR
Finalmente, tras todo el trabajo anterior, ya estamos en disposición de trabajar desde R sobre un cluster de máquinas, aprovechando todos sus recursos combinados desde una línea de comandos interactiva que, desde el principio, era mi objetivo, ya que esto me abre las puertas a trabajar con cantidades de datos mucho mayores sin necesidad recurrir a Java sobre Hadoop que, hasta el momento, es la combinación más usual.
Una vez que desde R hemos obtenido un contexto Spark, mediante la función sparkR.init, podemos usar la API RDD (Resilient Distributed Dataset) de Spark como si fuesen listas distribuidas en R. Además de extender la funcionalidad de construcciones habituales de R, como la función lapply, el paquete SparkR aporta un importante número de nuevas funciones como podemos ver en la ayuda electrónica.
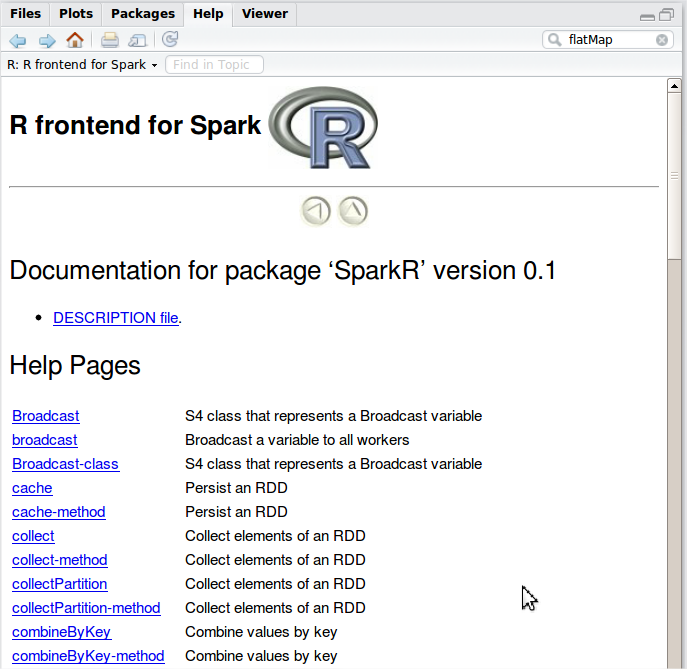
También tenemos a nuestra disposición múltiples ejemplos en el directorio examples del paquete SparkR, listos para ser cargados y ejecutados tal y como se puede ver en la imagen inferior.
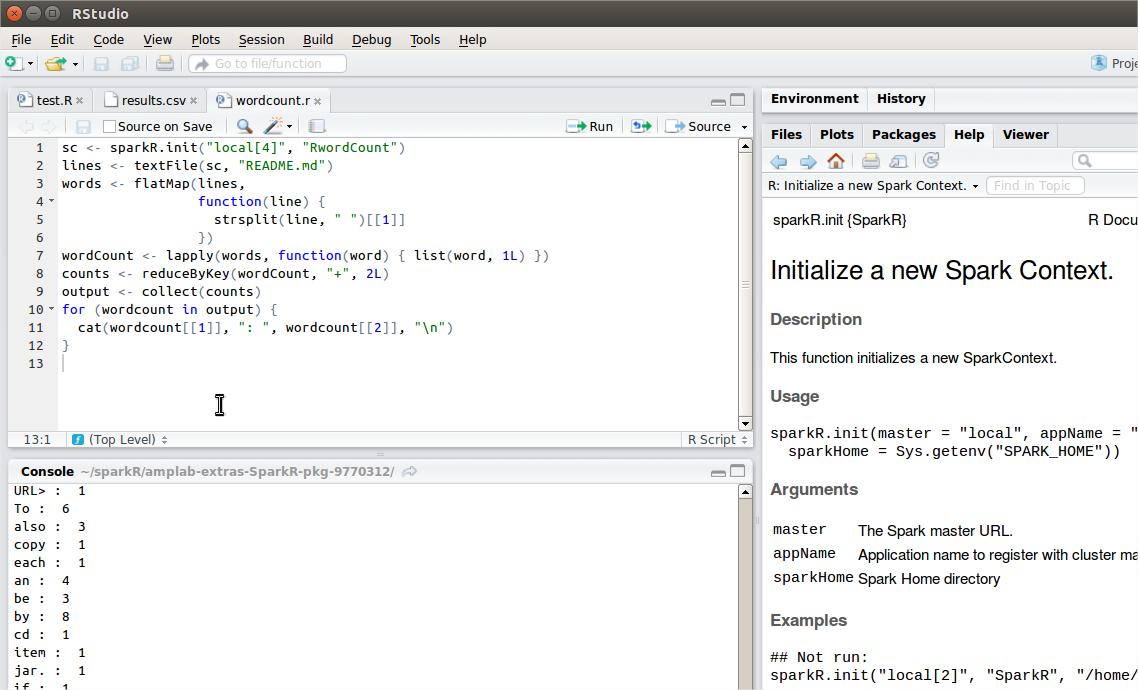
En cualquier momento podemos abrir el navegador y acceder a la consola de Spark que nos permite comprobar los trabajos que hay en curso y terminados, los recursos que se están utilizando en cada una de las máquinas, etc.
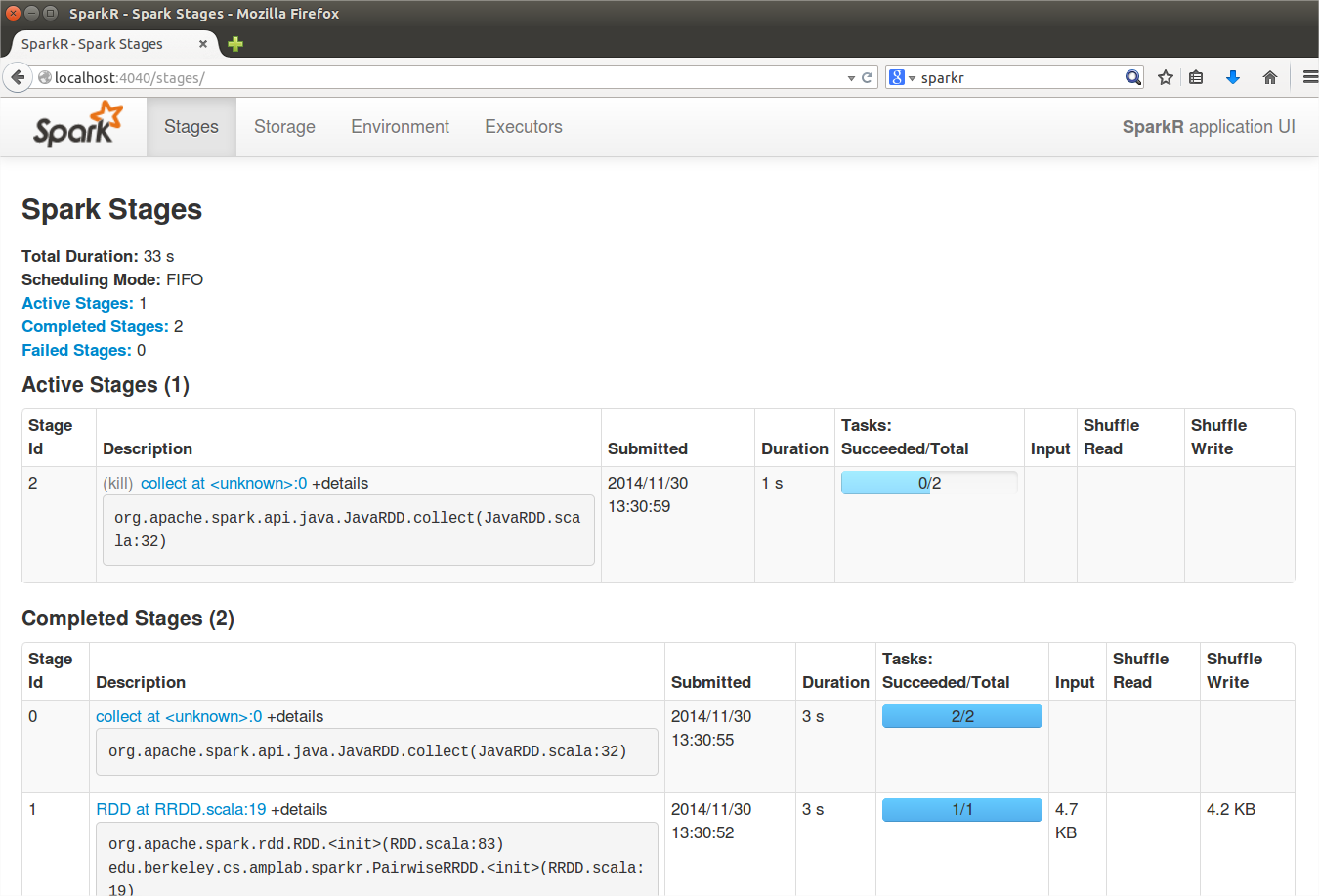
SparkR se encuentra aun en una fase muy preliminar. Es de esperar que en el futuro vayan extendiéndose sus capacidades y permita aprovechar todavía más las funciones de Spark, quizá incluyendo el acceso a librerías de machine learning como MLib. Queda mucho trabajo por delante.